- 科研进展
-
李富义教授课题组在基于生物语言模型的序列模 型挖掘研究上取得新进展
作者: 来源: 发布日期:2024-01-08 浏览次数:近日,李富义教授课题组在基于生物语言模型的序列模体挖掘研究上取得新进展,在《IEEE Journal of Biomedical and Health Informatics》(中科院工程技术大类一区,TOP期刊)发表了题为“PLANNER: a multi-scale deep language model for the origins of replication site prediction”的论文。该论文提出了基于生物语言模型真核生物复制起始位点预测算法模型PLANNER,融合不同尺度序列模体的全局上下文词嵌入,提升了真核生物复制起始位点预测精度,并结合深度学习可解释性技术,挖掘了真核生物复制起始位点的序列模体。课题组在读研究生王聪为论文第一作者,李富义教授为论文通讯作者。
为捕获真核生物复制起始位点的长距离DNA序列模体的依赖关系,该研究基于生物序列语言模型DNABERT,分别探讨了融合不同尺度的DNA序列全局上下文词嵌入方法在提升模型性能方面的效果,结果表明该方法显著提升了模型性能,相较于现有的基于传统机器学习算法和LSTM算法的模型,本研究提出的模型在精度和鲁棒性等方面取得了更优异的表现。PLANNER利用自注意力机制增强了模型的可解释性,并能挖掘出真核生物复制起始位点的重要序列模体,PLANNER与传统方法STREME挖掘出的序列模体具有很高的相似度,体现出PLANNER的序列模体挖掘能力。此外,为方便研究人员使用,该研究还开发了用于真核生物复制起始位点识别的在线预测平台(http://planner.unimelb-biotools.cloud.edu.au/),该平台自发布(2023年10月)至今已为来自全世界19个国家和地区的研究提供345次计算服务。
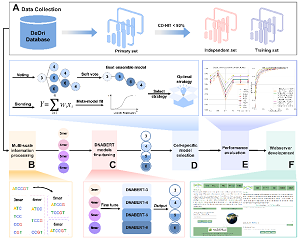
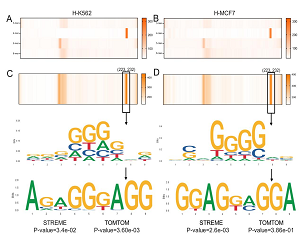
图1 PLANNER框架图 图2 序列模 型挖掘图
本研究得到国家重点研发计划(2022YFF1000100)、国家自然科学基金(62202388)、秦创原创新创业人才项目(QCYRCXM-2022-230)和太阳成集团tyc138高层次人才项目的资助。
原文链接:https://ieeexplore.ieee.org/document/10380693